こんにちは、データサイエンティストのたぬ(@tanuhack)です!
母分散が未知で、より現実的な正規分布の母平均の検定は、1標本のt検定と呼ばています。
これは『t検定統計量』と呼ばれるを検定統計量として使い、自由度n-1の『t分布』に従わせ、ビジネス上では主にA/Bテストの検証に用いられることが多いです。
数式が複雑で面倒な計算も、Pythonならちょちょいのちょい。
ということで今回は、Pythonを使って1標本t検定(両側/片側検定)を行う方法を紹介します!
目次
Pythonで実装する1標本t検定の流れ
- 仮説を立てる
- 有意水準を決める
- 検定統計量を計算
- p値を計算
- p値が有意水準より大きい:帰無仮説を採用
- p値が有意水準より小さい:帰無仮説を棄却
仮説の設定
あるカフェのコーヒー豆を15個サンプリングし、その平均が200gであるかどうか検定したい。
測定した15個のコーヒー豆の重量が次の通りである場合、検定の結果はどのようになるか答えよ。なお、有意水準は0.05とする。
210.9, 195.4, 202.1 , 211.3, 195.5, 212.9, 210.9, 198.3, 202.1, 215.6, 204.7, 212.2, 200.7, 206.1, 195.8
- 帰無仮説
:コーヒー豆の容量は200gである
- 対立仮説
:コーヒー豆の容量は200gではない
- 帰無仮説
:コーヒー豆の容量は200gである
- 対立仮説
:コーヒー豆の容量は200gより多い
Pythonで1標本t検定(両側/片側検定)を行う
Pythonで1標本t検定を行うためには、SciPyのscipy.stats.ttest_1sampメソッドを使います。1sampは1標本の略だと思われ。scipy.stats.ttest_1samp|公式サイト(英語)
scipy.stats.ttest_1sampメソッドを使えば、両側検定だろうが片側検定だろうが、一発で分かるので重宝しています。
t, p = sp.stats.ttest_1samp(a, popmean)| 必須オプション | 意味 |
|---|---|
| a | 数字の配列(Series、1d-array、list) |
| popmean | 帰無仮説で立てる母平均 |
ttest_1sampメソッドを使うと、2つの値(t値, p値)が格納されたタプルが返されるので、t検定で判断すべき数字は後者になります。
import numpy as np
import scipy as sp
coffee = np.array([
210.9, 195.4, 202.1 , 211.3, 195.5,
212.9, 210.9, 198.3, 202.1, 215.6,
204.7, 212.2, 200.7, 206.1, 195.8
])
t, p = sp.stats.ttest_1samp(coffee, popmean=200)
print('母平均が200のt値:'+str(t))
print('母平均が200である確率(p値):'+str(p))母平均が200のt値:2.751076959309973
母平均が200である確率(p値):0.015611934395473872
今回の場合、p値の値が1.5%だったので、両側検定でも片側検定でも共に帰無仮説が棄却され、対立仮説
が成り立ちます。
- 両側検定で帰無仮説を棄却させるには、p値が2.5%以下であれば良い
- 片側検定で帰無仮説を棄却させるには、p値が5%以下であれば良い
(余談ですが、片側検定の方が帰無仮説を棄却させやすいです)
1標本t分布をグラフで直感的に理解する
SciPyのscipy.stats.ttest_1sampメソッドで簡単に1標本t検定で使用するp値が得られました。
でも、このままだと、直感的にイメージが付きにくいと思いませんか??
ということで、先程得られたt値とp値を使って、matplotlibを使ってt分布を描画し、視覚的に帰無仮説を棄却できているのか確認してみましょう。
両側検定の場合
import numpy as np
import scipy as sp
from matplotlib import pyplot as plt
coffee = np.array([
210.9, 195.4, 202.1 , 211.3, 195.5,
212.9, 210.9, 198.3, 202.1, 215.6,
204.7, 212.2, 200.7, 206.1, 195.8
])
# 検定統計量
n = len(coffee) # サンプル数
k = n-1 # 自由度
u_var = np.var(coffee, ddof=1) # 不偏分散
s_mean = np.mean(coffee) # 標本平均
# t値とp値を求める
t, p = sp.stats.ttest_1samp(coffee, popmean=200)
# ここからグラフ描画
############################
x = np.linspace(-6, 6, 1000)
fig,ax = plt.subplots(1,1)
# t分布を描画する
ax.plot(x, sp.stats.t.pdf(x, k), linestyle='-', label='k='+str(k))
# t分布に今回の確率分布を表示させる
ax.plot(t, p, 'x', color='red', markersize=7, markeredgewidth=2, alpha=0.8, label='experiment')
# t分布の95%信頼区間から外れた領域を描画する
bottom, up = sp.stats.norm.interval(alpha=0.95, loc=0, scale=1)
plt.fill_between(x, sp.stats.t.pdf(x, k), 0, where=(x>=up)|(x<=bottom), facecolor='black', alpha=0.1)
plt.xlim(-6, 6)
plt.ylim(0, 0.4)
plt.legend()
plt.show()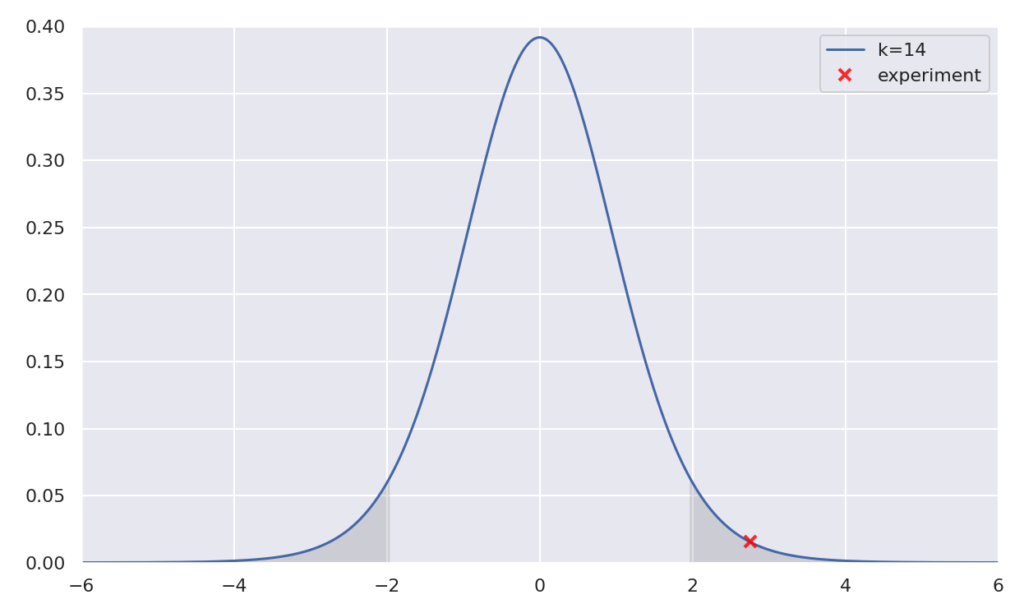
グラフを見ての通り、ちゃんと95%信頼区間以外に存在しているので、帰無仮説が棄却され、対立仮説
が成り立ちます。
片側検定(上側)の場合
import numpy as np
import scipy as sp
from matplotlib import pyplot as plt
coffee = np.array([
210.9, 195.4, 202.1 , 211.3, 195.5,
212.9, 210.9, 198.3, 202.1, 215.6,
204.7, 212.2, 200.7, 206.1, 195.8
])
# 検定統計量
n = len(coffee) # サンプル数
k = n-1 # 自由度
u_var = np.var(coffee, ddof=1) # 不偏分散
s_mean = np.mean(coffee) # 標本平均
# t値とp値を求める
t, p = sp.stats.ttest_1samp(coffee, popmean=200)
# ここからグラフ描画
############################
x = np.linspace(-6, 6, 1000)
fig,ax = plt.subplots(1,1)
# t分布を描画する
ax.plot(x, sp.stats.t.pdf(x, k), linestyle='-', label='k='+str(k))
# t分布に今回の確率分布を表示させる
ax.plot(t, p, 'x', color='red', markersize=7, markeredgewidth=2, alpha=0.8, label='experiment')
# t分布の90%信頼区間から外れた領域を描画する
bottom, up = sp.stats.norm.interval(alpha=0.90, loc=0, scale=1)
plt.fill_between(x, sp.stats.t.pdf(x, k), 0, where=(x>=up), facecolor='black', alpha=0.1)
plt.xlim(-6, 6)
plt.ylim(0, 0.4)
plt.legend()
plt.show()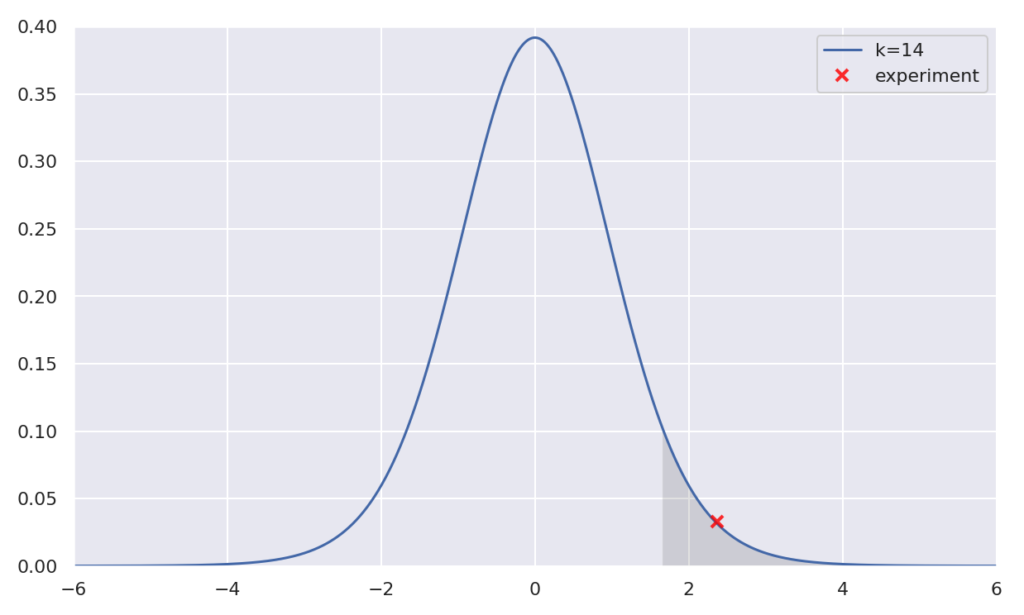
こちらもグラフを見ての通り、ちゃんと95%信頼区間以外に存在しているので、帰無仮説が棄却され、対立仮説
が成り立ちます。
片側検定の下側を確認したい場合はプログラムを以下のように修正して下さい。
plt.fill_between(x, sp.stats.t.pdf(x, k), 0, where=(x<=bottom), facecolor='black', alpha=0.1)


