こんにちは、データサイエンティストのたぬ(@tanuhack)です!
『母平均が分からないのに母分散だけは分かっている』状況は、現実世界ではほぼありえません。
統計学では、サンプルから得た情報で『母平均と母分散がともに未知の正規分布に従う』として、サンプルサイズに応じて、『t分布』または『正規分布』を使い、母平均の区間推定を行ないます。
- 30以下:t分布
- 30以上:t分布または正規分布
ですが結論から言うと、母平均の区間推定はt分布だけで大丈夫です。
それは、サンプルサイズが30を超えたあたりからt分布が正規分布とほぼ一致するからです。
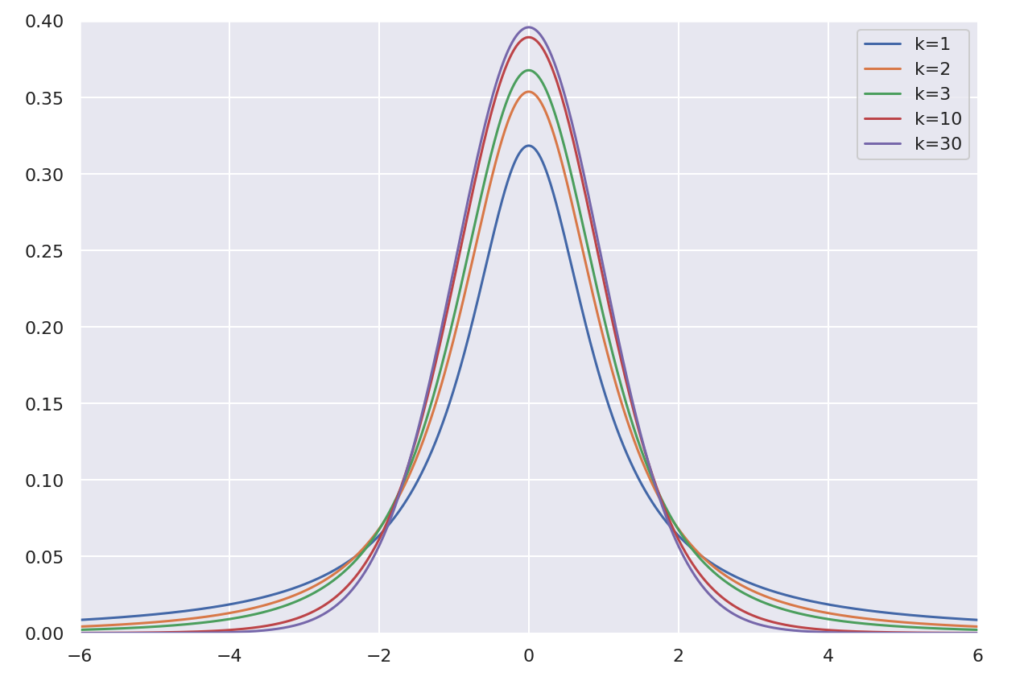
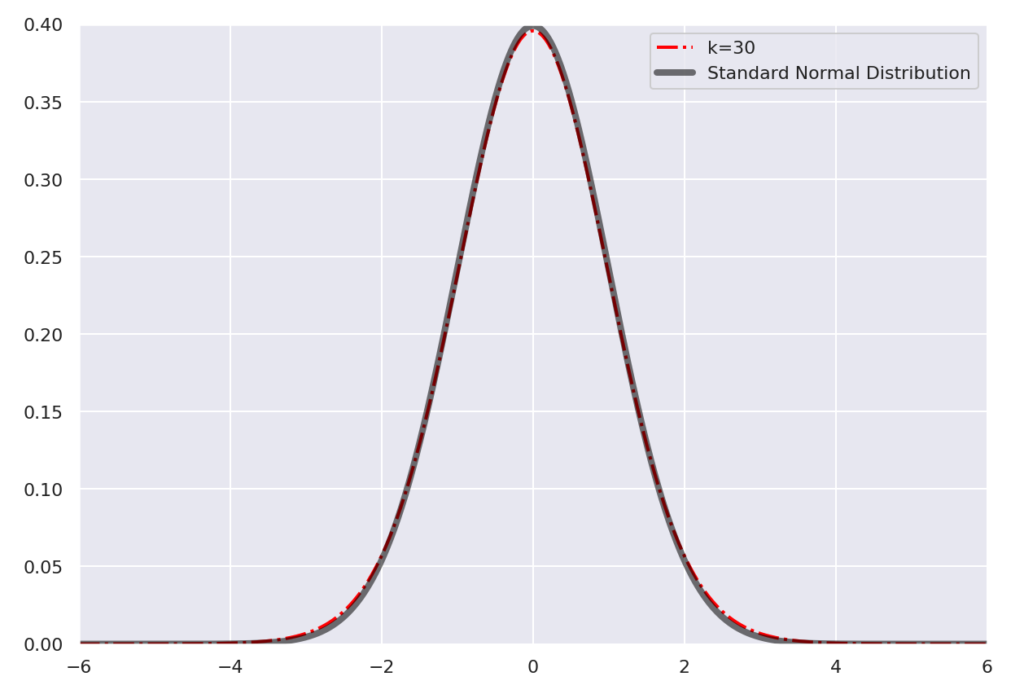
繰り返しになりますが以上のことから、統計学ではサンプルから得た情報で『母平均と母分散がともに未知の正規分布に従う』としてt分布を使い、母平均の区間推定を行ないます。
t分布で母平均の95%信頼区間を推定する
Pythonを使って、t分布で母平均の95%信頼区間を推定するためには、SciPyの
scipy.stats.t.interval関数を使います。
bottom, up = scipy.stats.t.interval(alpha=A, loc=B, scale=C, df=D)| interval関数の必須オプション | 意味 | 記号 |
|---|---|---|
alpha |
信頼区間(0.95=95%、0.99=99%など) | |
loc |
標本平均 | |
scale |
標準誤差(推定量の標準偏差) | |
df |
自由度(サンプルサイズ-1) |
1つだけ注意しなければいけないのが、scaleオプションでは、標本標準偏差ではなく推定量の標準偏差である標準誤差(不偏分散
をサンプルサイズ
で割った平方根)を使用しなければいけません。
以下、Pythonでt分布で母平均の95%信頼区間を推定する流れです。
- ライブラリのインポート、分析で使うデータをセットする
- 不偏分散
と標本平均
を求める
- t分布の自由度を求める
- t分布の両側2.5%で区間推定する
順に紹介していきます!
ライブラリとデータの準備
まず、必要なライブラリのインポートと、今回使うアヤメの統計データをセットします。
STEP1:ライブラリのインポート
import numpy as np
import pandas as pd
import scipy as sp
import seaborn as sns # ==> pip install seaborn
%precision 3STEP2:アヤメの統計データをセット
データセットは、Rでお馴染みの『アヤメの統計データ』をPandasのDataFrameに格納させます。
アヤメのデータセットは、seabornに予め用意されているのでそれを使います。ありがたや〜。
iris = sns.load_dataset('iris')
# type(iris) ==> pandas.core.frame.DataFrame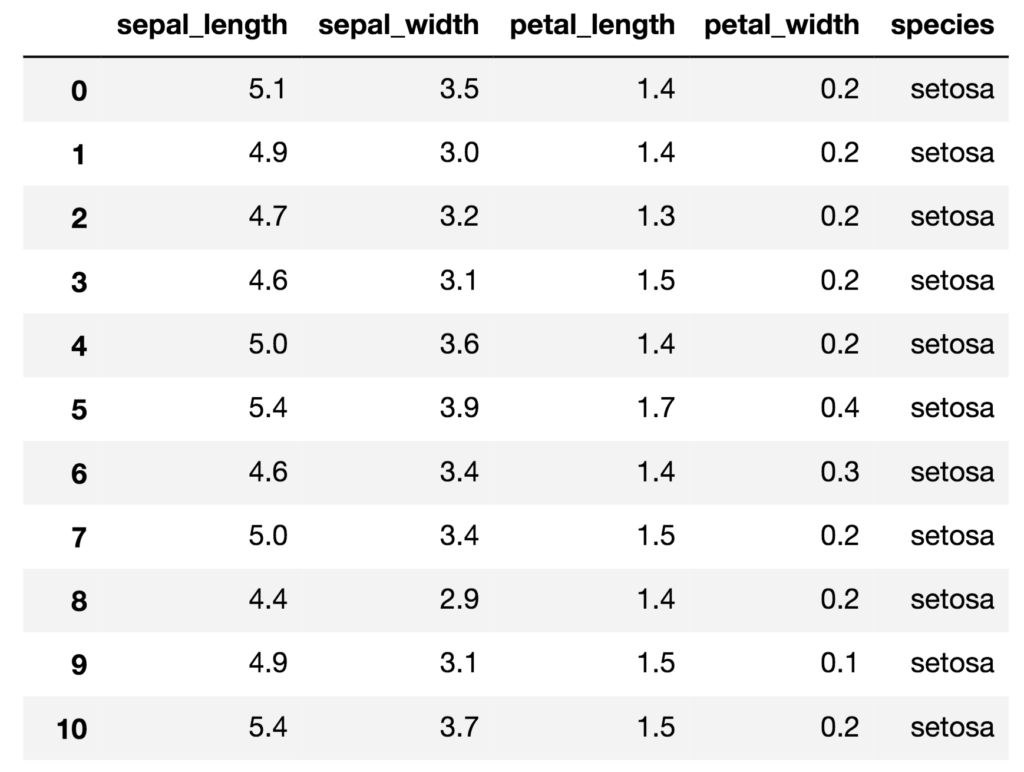
| レコード数 | 150 |
|---|---|
| カラム数 | 5 |
| カラム名 | 説明 | データの尺度名 |
|---|---|---|
| sepal_length | がく片の長さ:4.3~7.9(cm) | 間隔尺度(量的変量) |
| sepal_width | がく片の幅:2.0~4.4(cm) | 間隔尺度(量的変量) |
| petal_length | 花びらの長さ:1.3~6.9(cm) | 間隔尺度(量的変量) |
| petal_width | 花びらの幅:0.1~2.5(cm) | 間隔尺度(量的変量) |
| species | アヤメの種類(setosa, versicolor, virginica) | 名義尺度(質的変量) |
irisデータから、アヤメのsetosa種のpetal_length(花びらの長さ)を20個ランダムで抽出して、母平均の95%信頼区間を算出する
STEP3:setosa種のpetal_length(花びらの長さ)を20個ランダムサンプリングする
# アヤメのsetosaから花びらの長さ(petal_length)を20個ランダムで抽出する
np.random.seed(111) # ランダムサンプリングの値を固定
setosa_petal_length = np.array(iris[iris['species']=='setosa']['petal_length'].sample(20))
setosa_petal_length不偏分散と標本平均を求める
PythonのNumPyではnumpy.var(date, ddof=1)で標本の不偏分散を算出します。(ddof=0は標本分散)
# 不偏分散(Unbiased Variance)と標本平均(Sample Mean)を求める
u_var = np.var(setosa_petal_length, ddof=1)
s_mean = np.mean(setosa_petal_length)
u_var, s_mean抽出した標本の平均値は、となり、不偏分散は
となります。
t分布の自由度を求める
t分布では『サンプル数-1』が自由度になります。
# 使用するt分布の自由度(degree of freedom)を決める
# サンプルサイズが20であることから自由度が20-1=19のt分布を用いる
deg_of_freedom = len(setosa_sepal_length)-1
deg_of_freedom今回の区間推定で用いるt分布の自由度は、20-1で19となります。
t分布の両側2.5%で区間推定する
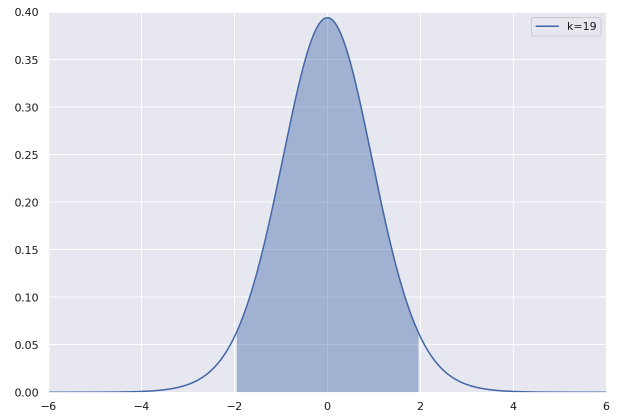
# t分布の両側2.5%で区間推定する
bottom, up = sp.stats.t.interval(
alpha=0.95, # 信頼区間
loc=s_mean, # 標本平均
scale=np.sqrt(u_var/len(setosa_petal_length)), # 標準誤差(推定量の標準偏差)
df=deg_of_freedom # 自由度
)
print('母平均の95%信頼区間: {:.2f} < μ < {:.2f}'.format(bottom, up))したがって、完全未知なアヤメのsetosa種の花びらの長さの平均値(定数)は、95%の確率でおおよそ1.41cmから1.53cmだろうと推定することができました。
さいごに
今回は、t分布で母平均の95%信頼区間を推定するために、PythonのSciPyライブラリの
scipy.stats.t.interval関数を使って算出しました。
算出された母平均の推定区間を狭めたい場合は、次のどちらかを行えばOKです。
- 信頼係数を小さくする(95%→90%など)
- サンプルサイズを大きくする(20個→100個など)
ただ、信頼係数を下げるのはあまり現実的だとは言えないので、よほどのことが無い限りはサンプルサイズを大きくすることを意識しましょう。
それでは



